摘要: 一项新的研究发现,“随机DNA”在单细胞真菌酵母中自然活跃。
一项新的研究发现,“随机DNA”在单细胞真菌酵母中自然活跃,而在哺乳动物细胞中,这种DNA在其自然状态下被关闭,尽管它们在10亿年前有共同的祖先,并且具有相同的基本分子机制。
这项新发现围绕着DNA遗传指令首先转化为一种名为RNA的相关物质,然后转化为构成身体结构和信号的蛋白质的过程展开。在酵母、小鼠和人类中,基因表达的第一步,转录,是DNA分子“字母”(核碱基)在一个方向上被读取时进行的。虽然80%的人类基因组——我们细胞中的全套DNA,被积极地解码成RNA,但实际上只有不到2%的基因编码指导蛋白质的构建。
基因组学中一个长期存在的谜团是,所有这些非基因相关的转录是如何完成的。它只是噪音,是进化的副作用,还是有它的功能?
纽约大学朗格尼健康中心(NYU Langone Health)的一个研究小组试图通过创造一个巨大的合成基因来回答这个问题,该基因的DNA编码与自然母体的顺序相反。然后,他们将合成基因放入酵母和小鼠干细胞中,观察它们的转录水平。
这项新研究发表在3月6日的《自然》杂志上,揭示了酵母的遗传系统是这样设置的,几乎所有的基因都在持续转录,而在哺乳动物细胞中,同样的“默认状态”是转录被关闭。
这项发表在《自然通讯》杂志上的研究不仅揭示了IncREST是应激反应的关键组成部分,而且还可能是对抗各种癌症的有效治疗靶点。
 图1 反向合成序列揭示默认的基因组状态
图1 反向合成序列揭示默认的基因组状态有趣的是,该研究的作者说,代码的反向顺序意味着酵母和哺乳动物细胞中进化的所有开启或关闭转录的机制都不存在,因为反向代码是无意义的。然而,就像镜像一样,反向密码反映了自然密码中的一些基本模式,包括DNA字母出现的频率、它们靠近的位置以及它们重复的频率。由于反向代码有10万个分子字母长,研究小组发现它随机包含了许多以前未知的代码的小片段,这些代码可能更频繁地开始转录,并在哺乳动物细胞中停止转录。
“了解不同物种之间的默认转录差异将有助于我们更好地了解遗传密码的哪些部分具有功能,哪些是进化的意外,这反过来又有望指导酵母的工程,制造新药,或创造新的基因疗法,甚至帮助我们找到隐藏在庞大密码中的新基因。”通讯作者 Jef Boeke博士说。
这项研究也为以下理论提供了依据:酵母非常活跃的转录状态是固定的,因此外来DNA很可能被转录成RNA。外来DNA很少被注入酵母,比如病毒在复制自身时注入酵母。如果RNA构建了一种具有有益功能的蛋白质,那么这种编码将作为一种新基因被进化保存下来。与酵母中的单细胞生物不同,哺乳动物细胞作为由数百万个协同细胞组成的身体的一部分,每当细胞遇到病毒时,它们就不那么自由地吸收新的DNA。酵母中的单细胞生物可以提供有风险的新基因,从而加速进化。许多监管机制保护微妙平衡的代码。
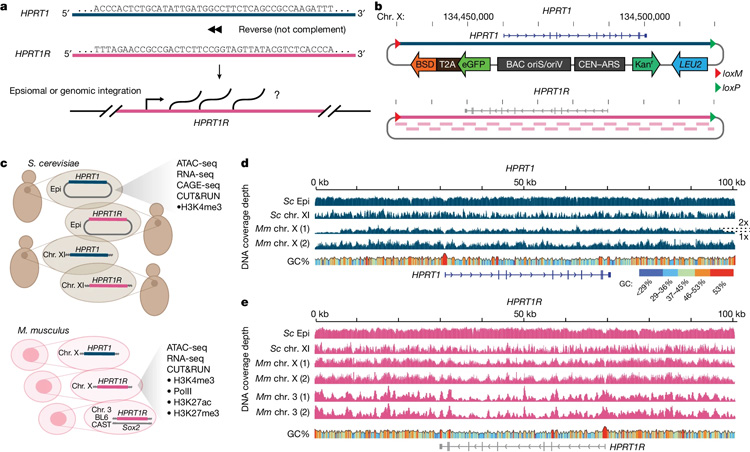 图2 合成HPRT1和HPRT1R的设计和构建
图2 合成HPRT1和HPRT1R的设计和构建大DNA
这项新研究必须考虑到DNA链的大小,人类基因组中包含30亿个“字母”,有些基因长达200万个字母。虽然著名的技术可以一个字母一个字母地进行改变,但如果研究人员从零开始构建DNA,将大量预先组装的代码交换到一个细胞中,以取代自然对应的细胞,对一些工程任务进行广泛的改变,效率会更高。
由于人类基因非常复杂,Boeke的实验室首先在酵母中开发了“genome writing”方法,将其应用于哺乳动物的遗传密码。该研究的作者使用酵母细胞在一个步骤中组装长DNA序列,然后将它们放入小鼠胚胎干细胞中。
在最新研究中,研究小组通过以反向编码顺序引入合成的101千碱基工程DNA -人类基因次黄嘌呤磷酸核糖基转移酶1 (HPRT1),解决了转录在进化过程中如何普遍存在的问题。他们观察到该基因在酵母中的广泛活性,尽管缺乏启动子的无意义代码,即进化为转录开始信号的DNA片段。
此外,研究小组还发现了反向编码中的小序列,腺苷和胸腺嘧啶构建块的重复延伸,已知可被转录因子识别,转录因子是与DNA结合以启动转录的蛋白质。作者说,只有5到15个碱基长,这样的序列很容易随机出现,可能部分解释了酵母非常活跃的默认状态。
相反,将相同的反向编码插入小鼠胚胎干细胞的基因组中,并没有引起广泛的转录。在这种情况下,即使进化的CpG二核苷酸(已知主动关闭(沉默)基因)在反向编码中不起作用,转录也受到抑制。研究小组推测,哺乳动物基因组中的其他基本元素可能比酵母更严格地限制转录,可能是通过直接招募一种已知的沉默基因的蛋白质组(多梳复合体)来实现的。
第一作者Brendan Camellato是Boeke实验室的研究生,他说:“我们越接近将‘基因组价值’的无意义DNA引入活细胞,就越能更好地将其与实际的进化基因组进行比较。这可能会引领我们进入工程细胞疗法的新前沿,因为植入更长的合成dna的能力使我们能够更好地了解基因组能够耐受的插入物,也许还能包含一个或多个更大、完整的工程基因。”
参考资料
[1] Synthetic reversed sequences reveal default genomic states